Rで構造方程式モデリング(共分散構造分析)を実行する場合、semパッケージかlavaanパッケージを使うことが多いです。
https://socialsciences.mcmaster.ca/jfox/Misc/sem/index.htmlsocialsciences.mcmaster.ca
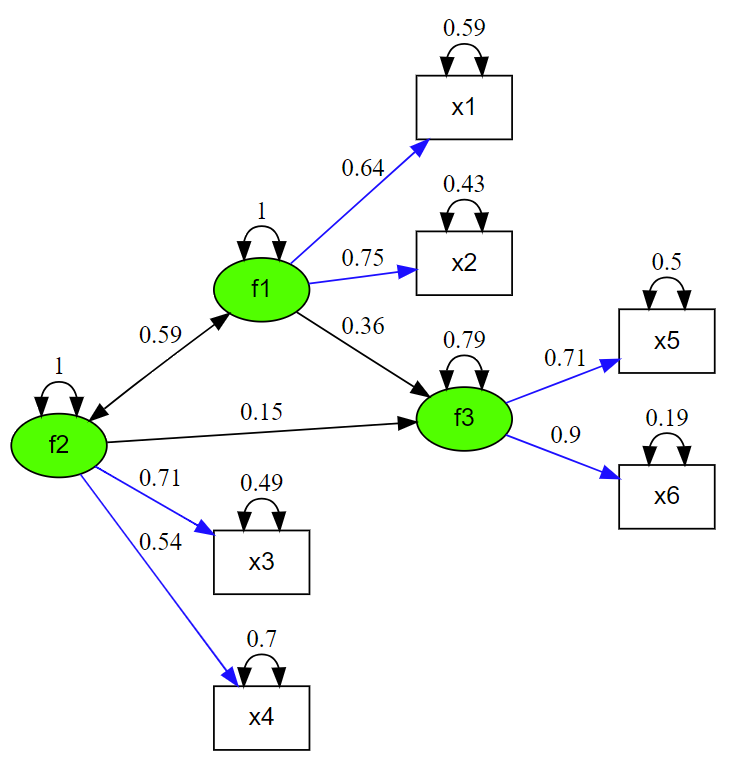
構造方程式モデリングを実施したらパス図が書きたくなります。
semパッケージやlavaanパッケージで作ったモデルからパス図を描くパッケージとしてsemPlotパッケージがありますが、なんかしっくりこない。
http://sachaepskamp.com/documentation/semPlot/semPaths.htmlsachaepskamp.com
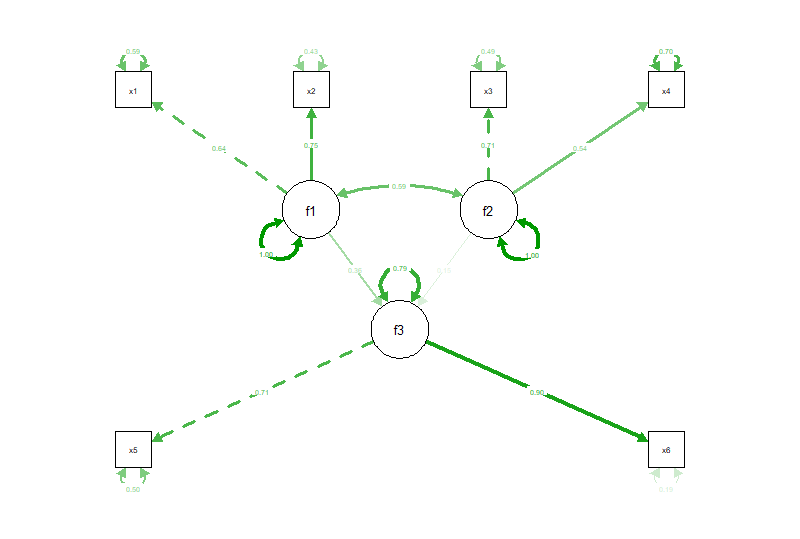
そこで自分で lavaan のモデルからDiagrammeRを利用してキレイなパス図を描く関数を作りました。
rich-iannone.github.io
my_lavaan_plot <- function(fit, standardized = TRUE) { require(lavaan) require(stringr) require(dplyr) require(DiagrammeR) #描画に必要なパラメータをdfとして取り出す pars <- parameterEstimates(fit, standardized = standardized) # エッジ(パス)の設定、"=~"を抜き出しpaseteして因子負荷量 temp01 <- dplyr::filter(pars, op == "=~") temp02 <- str_c(' \"', temp01$lhs,'\"', ' -> ','\"', temp01$rhs, '\"', '[label=\"', round(temp01$std.all,2), '\" color=blue penwidth=1.001];') # "~"を抜き出しpaseteして因子間回帰 temp31 <- dplyr::filter(pars, op == "~") temp32 <- str_c(' \"', temp31$rhs,'\"', ' -> ','\"', temp31$lhs, '\"', '[label=\"', round(temp31$std.all,2), '\" color=black penwidth=1.001];') # "~~"抜き出す temp11 <- dplyr::filter(pars, op == "~~") temp12 <- str_c(' \"', temp11$rhs,'\"', ' -> ','\"', temp11$lhs, '\"', '[label=\"', round(temp11$std.all,2), '\" color=gray penwidth=1.001, dir = both];') # ノードの設定 観測変数と潜在因子 temp00 <- inspect(fit)$lambda test.f <- colnames(temp00) #潜在因子 test.v <- rownames(temp00) #観測変数 temp21 <- str_c(' \"', test.f,'\" [fontname=\"Helvetica\" fontsize=14 fillcolor = green shape=ellipse style=filled];') temp22 <- str_c(' \"', test.v,'\" [fontname=\"Helvetica\" fontsize=14 fillcolor=\"transparent\" shape=box style=filled];') tempALL <- c('digraph \"res.s\" {', ' layout = dot;', #dot, fdp ' rankdir=LR;', ' size=\"8,8\";', temp21, temp22, temp02, temp32, ' center=1;', temp12, '}') grViz(tempALL) }
使い方はlavaanで作ったモデルを食わせるだけ。
# テスト用のモデル # ヘッドスタート計画の相関係数行列 HS <- matrix(c(1.000, 0.484, 0.224, 0.268, 0.230, 0.265, 0.484, 1.000, 0.342, 0.215, 0.215, 0.295, 0.224, 0.342, 1.000, 0.387, 0.196, 0.234, 0.268, 0.215, 0.387, 1.000, 0.115, 0.162, 0.230, 0.215, 0.196, 0.115, 1.000, 0.635, 0.265, 0.295, 0.234, 0.162, 0.635, 1.000), 6) colnames(HS) <- rownames(HS) <- paste("x", 1:6, sep="") HS.mdl.2 <- ' # 測定方程式 f1 =~ x1 + x2 f2 =~ x3 + x4 f3 =~ x5 + x6 # 構造方程式 f3 ~ f1 + f2 ' fit <- sem(HS.mdl.2, sample.cov=HS, sample.nobs=1800) summary(fit, fit.measures=TRUE, standardized=TRUE) my_lavaan_plot(fit)